Charger les données
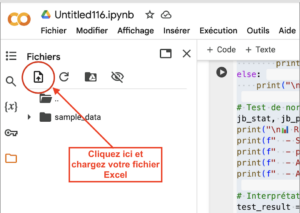
Trouvez le chemin d’accès aux données
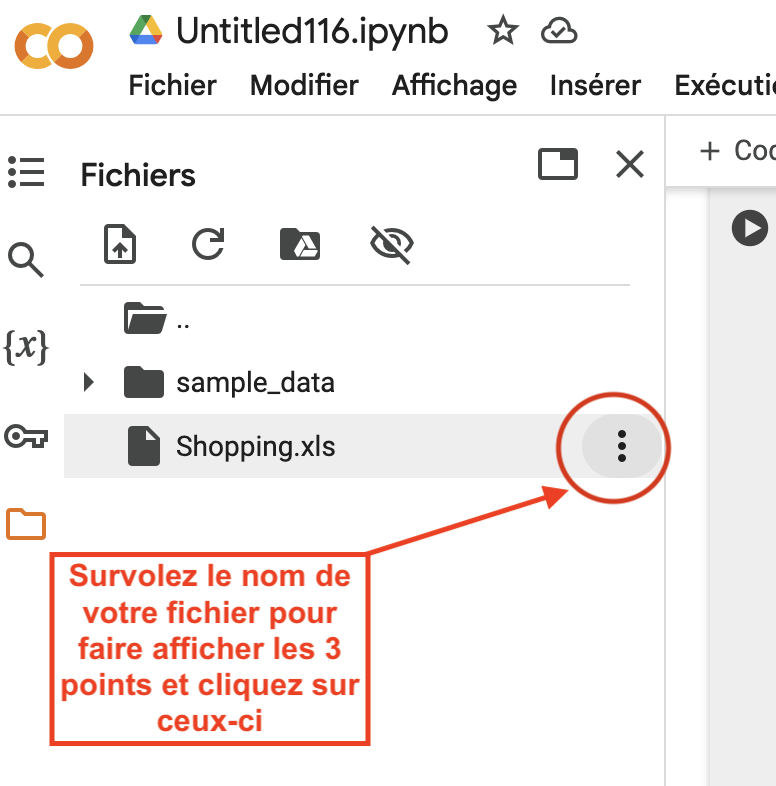
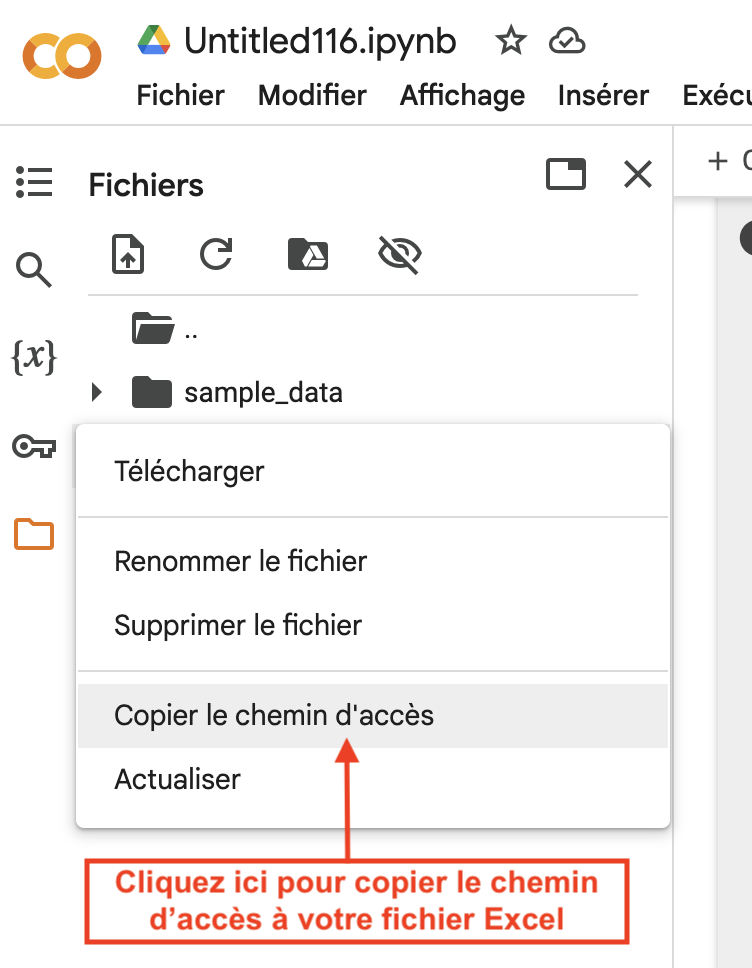
Copiez le chemin d’accès aux données
Chargez les bibliothèques nécessaires
Copiez le code ci-dessous et collez-le sous Google Colab :
import pandas as pd
# Demander à l'utilisateur d'entrer le chemin du fichier
file_path = input("Veuillez coller le chemin complet du fichier XLS : ")
# Charger le fichier XLS dans un DataFrame
data_original = pd.read_excel(file_path)
# Afficher le contenu du DataFrame
data_original
Installer les bibliothèques nécessaires
Copiez le code ci-dessous et collez-le sous Google Colab :
!pip install factor_analyzer
!pip install --upgrade scikit-learn
!pip install --upgrade --no-deps scikit-learn
!pip install --upgrade imbalanced-learn xgboost
Procéder à l’Analyse en Composantes Principales sans rotation
Copiez le code ci-dessous et collez-le sous Google Colab :
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
from sklearn.preprocessing import StandardScaler
from factor_analyzer import FactorAnalyzer
from factor_analyzer.factor_analyzer import calculate_kmo, calculate_bartlett_sphericity
from ipywidgets import interactive
import ipywidgets as widgets
# Fonction pour calculer l'alpha de Cronbach
def cronbach_alpha(df):
if len(df.columns) > 1:
k = df.shape[1]
variances = df.var(axis=0, ddof=1)
total_variance = df.sum(axis=1).var(ddof=1)
return (k / (k - 1)) * (1 - (variances.sum() / total_variance))
else:
print("⚠️ Pas assez de variables pour calculer l'alpha sans item.")
# Chargement des données (supposées déjà chargées dans data_original)
def select_columns(data):
print("\nColonnes disponibles :", list(data.columns))
selected_columns = input("Entrez les noms des colonnes pour l'ACP (séparées par une virgule) : ").strip().split(',')
selected_columns = [col.strip() for col in selected_columns]
if not all(col in data.columns for col in selected_columns):
print("Erreur : certaines colonnes n'existent pas. Réessayez.")
return select_columns(data)
return selected_columns
# Étape 1 : Sélection des colonnes pour l'ACP
selected_columns = select_columns(data_original)
df = data_original[selected_columns]
# Étape 2 : Normalisation des données
scaler = StandardScaler()
data_scaled = scaler.fit_transform(df)
# Étape 3 : Test KMO et Bartlett
kmo_all, kmo_model = calculate_kmo(df)
chi_square_value, p_value = calculate_bartlett_sphericity(df)
print(f"\nKMO global : {kmo_model:.3f}")
print(f"Test de Bartlett : Chi² = {chi_square_value:.3f}, p = {p_value:.3f}")
# Étape 4 : Communalités
# Affichage des communalités
fa_temp = FactorAnalyzer(rotation=None, method='principal', is_corr_matrix=False)
fa_temp.fit(data_scaled)
eigenvalues, _ = fa_temp.get_eigenvalues()
n_factors = sum(eigenvalues > 1)
print(f"Nombre de composantes retenues (valeurs propres > 1) : {n_factors}")
fa_temp = FactorAnalyzer(rotation=None, method='principal', n_factors=n_factors,is_corr_matrix=False)
fa_temp.fit(data_scaled)
communalities = pd.DataFrame({
'Variable': df.columns,
'Initial': [1.000] * len(df.columns), # Les valeurs initiales sont toujours 1
'Communalité': fa_temp.get_communalities()
}).round(3)
print("\nCommunalités des variables:")
print(communalities)
# Étape 5 : Détermination du nombre de facteurs
eigenvalues, _ = fa_temp.get_eigenvalues()
n_factors = sum(eigenvalues > 1)
print(f"Nombre de composantes retenues (valeurs propres > 1) : {n_factors}")
# Tableau des variances expliquées
explained_variance_ratio = eigenvalues / np.sum(eigenvalues)
cumulative_variance = np.cumsum(explained_variance_ratio)
variance_table = pd.DataFrame({
'Composante': [f"Composante {i+1}" for i in range(len(eigenvalues))],
'Valeurs propres': eigenvalues,
'Variance expliquée (%)': explained_variance_ratio * 100,
'Variance expliquée cumulée (%)': cumulative_variance * 100
}).round(3)
display(variance_table)
# Graphique Scree Plot
plt.figure(figsize=(8, 6))
plt.plot(range(1, len(eigenvalues) + 1), eigenvalues, marker='o')
plt.title("Graphique des valeurs propres (Scree Plot)")
plt.xlabel("Composantes")
plt.ylabel("Valeurs propres")
plt.grid(True)
plt.show()
# Étape 6 : Choix du nombre de facteurs par l'utilisateur
# Demander à l'utilisateur le nombre de composantes à retenir au moment de la sauvegarde
n_factors = int(input("Entrez le nombre de composantes à retenir pour les scores factoriels : "))
# Étape 7 : Analyse factorielle avec le nombre de facteurs sélectionné
fa = FactorAnalyzer(rotation=None, method='principal', n_factors=n_factors)
fa.fit(data_scaled)
# Matrice des charges factorielles
loadings = fa.loadings_
components_matrix = pd.DataFrame(loadings, index=df.columns).round(3)
display(components_matrix)
# Étape 8 : Cercle des corrélations
if n_factors > 1:
fig, ax = plt.subplots(figsize=(6, 6))
ax.set_xlim(-1, 1)
ax.set_ylim(-1, 1)
ax.axhline(0, color='black', linestyle='-', linewidth=0.8)
ax.axvline(0, color='black', linestyle='-', linewidth=0.8)
ax.grid(True, linestyle='--', linewidth=0.5, alpha=0.7)
for i in range(loadings.shape[0]):
ax.scatter(loadings[i, 0], loadings[i, 1], color='b', alpha=0.5, s=50)
ax.text(loadings[i, 0], loadings[i, 1], df.columns[i], color='r', fontsize=8, ha='center')
ax.set_title("Cercle des corrélations", fontsize=10)
ax.set_xlabel("Composante 1")
ax.set_ylabel("Composante 2")
plt.show()
# Demander à l'utilisateur s'il souhaite une rotation
rotation_choice = input("Souhaitez-vous procéder à une rotation des variables ? (oui/non) : ").strip().lower()
if rotation_choice == "non":
fa = FactorAnalyzer(n_factors=n_factors, rotation=None, method='principal')
fa.fit(data_scaled)
factor_scores = fa.transform(data_scaled)
# Demander à l'utilisateur le nom des composantes
component_names = []
for i in range(n_factors):
name = input(f"Entrez le nom pour la composante {i+1} : ")
component_names.append(name)
components_matrix.columns = component_names
factor_scores_df = pd.DataFrame(factor_scores, columns=component_names)
# Affichage des scores factoriels
display(factor_scores_df)
# Calcul de l'alpha de cronbach
global_alpha = cronbach_alpha(df)
print(f"Alpha de Cronbach global : {global_alpha:.3f}")
alpha_sans_item = {col: cronbach_alpha(df.drop(columns=[col])) for col in selected_columns}
df_alpha = pd.DataFrame.from_dict(alpha_sans_item, orient='index', columns=['Alpha sans item']).round(3)
display(df_alpha)
############# ROTATION ##################@@
else:
rotation_types = ["varimax", "promax", "oblimin", "quartimax"]
print("Types de rotation disponibles :", rotation_types)
rotation_type = input("Veuillez entrer le type de rotation souhaité : ").strip().lower()
if rotation_type not in rotation_types:
print("Type de rotation non valide. Par défaut, 'varimax' sera utilisé.")
rotation_type = "varimax"
fa = FactorAnalyzer(n_factors=n_factors, rotation=rotation_type, method='principal')
fa.fit(data_scaled)
loadings = fa.loadings_
components_matrix = pd.DataFrame(loadings, index=df.columns).round(3)
# Affichage des scores factoriels
# display(factor_scores_df)
# Étape 8 : Cercle des corrélations
if n_factors > 1:
fig, ax = plt.subplots(figsize=(6, 6))
ax.set_xlim(-1, 1)
ax.set_ylim(-1, 1)
ax.axhline(0, color='black', linestyle='-', linewidth=0.8)
ax.axvline(0, color='black', linestyle='-', linewidth=0.8)
ax.grid(True, linestyle='--', linewidth=0.5, alpha=0.7)
for i in range(loadings.shape[0]):
ax.scatter(loadings[i, 0], loadings[i, 1], color='b', alpha=0.5, s=50)
ax.text(loadings[i, 0], loadings[i, 1], df.columns[i], color='r', fontsize=8, ha='center')
ax.set_title("Cercle des corrélations", fontsize=10)
ax.set_xlabel("Composante 1")
ax.set_ylabel("Composante 2")
plt.show()
# Demander à l'utilisateur le nom des composantes
component_names = []
for i in range(n_factors):
name = input(f"Entrez le nom pour la composante {i+1} : ")
component_names.append(name)
components_matrix.columns = component_names
factor_scores = fa.transform(data_scaled)
factor_scores_df = pd.DataFrame(factor_scores, columns=component_names)
# Affichage des scores factoriels
display(factor_scores_df)
global_alpha = cronbach_alpha(df)
print(f"Alpha de Cronbach global : {global_alpha:.3f}")
alpha_sans_item = {col: cronbach_alpha(df.drop(columns=[col])) for col in selected_columns}
df_alpha = pd.DataFrame.from_dict(alpha_sans_item, orient='index', columns=['Alpha sans item']).round(3)
display(df_alpha)
def generate_pca_summary(data_original, n_factors, fa, kmo_model, global_alpha, eigenvalues):
"""
Génère un tableau de synthèse des résultats de l'ACP si une seule dimension est retenue.
Args:
df (pd.DataFrame): Les données utilisées pour l'ACP.
n_factors (int): Nombre de facteurs extraits.
fa (FactorAnalyzer): Analyseur factoriel après ajustement des données.
kmo_model (float): Indice KMO global.
alpha_cronbach (float): Alpha de Cronbach de l'échelle.
eigenvalues (list): Valeurs propres des composantes.
Returns:
pd.DataFrame: Un tableau de synthèse des résultats.
"""
if n_factors == 1:
communalities = fa.get_communalities()
loadings = fa.loadings_
summary_data = []
for i, var in enumerate(df.columns):
summary_data.append([
var,
loadings[i][0],
"-", # Alpha sans item (à calculer si nécessaire)
communalities[i],
1.000 if i == 0 else round(np.corrcoef(df.iloc[:, 0], df.iloc[:, i])[0, 1], 3)
])
summary_data.append(["KMO", kmo_model, "-", "-", "-"])
summary_data.append(["α de l’échelle", global_alpha, "-", "-", "-"])
summary_data.append(["Valeur propre du facteur", eigenvalues[0], "-", "-", "-"])
summary_data.append(["Variance expliquée", round((eigenvalues[0] / sum(eigenvalues)) * 100, 3), "-", "-", "-"])
columns = ["Items", "Contributions factorielles", "α sans item", "Qualité Extraction", "Correl. PROP1"]
return pd.DataFrame(summary_data, columns=columns)
return None # Retourne None si plus d'un facteur est extrait
# Génération du tableau si une seule dimension est extraite
pca_summary = generate_pca_summary(data_original, n_factors, fa, kmo_model, global_alpha, eigenvalues)
# Affichage du tableau
if pca_summary is not None:
#import ace_tools as tools
#tools.display_dataframe_to_user(name="Tableau de Synthèse PCA", dataframe=pca_summary)
print(pca_summary)
from IPython.display import display
display(pca_summary)
# Demander à l'utilisateur s'il souhaite enregistrer les résultats
def save_results():
global data_original
data_original = pd.concat([data_original, factor_scores_df], axis=1)
output_file = "Analyse_Factorielle_Resultats.xlsx"
with pd.ExcelWriter(output_file) as writer:
factor_scores_df.to_excel(writer, sheet_name="Scores_Factoriels", index=False)
df_alpha.to_excel(writer, sheet_name="Alpha_Cronbach", index=True)
print(f"\nLes résultats ont été sauvegardés dans {output_file}.")
save_btn = widgets.Button(description="Enregistrer les résultats")
save_btn.on_click(lambda b: save_results())
display(save_btn)Procéder à la régression
Copiez le code ci-dessous et collez-le sous Google Colab :
import pandas as pd
import numpy as np
import statsmodels.api as sm
import matplotlib.pyplot as plt
import seaborn as sns
from sklearn.preprocessing import StandardScaler
from statsmodels.stats.outliers_influence import variance_inflation_factor
# Sélectionner les variables explicatives (X) et la variable dépendante (y)
y_var = input("Entrez le nom de la variable dépendante : ").strip()
X_vars = input("Entrez les noms des variables explicatives (séparés par une virgule) : ").strip().split(",")
# Vérification des variables
if y_var not in data_original.columns or any(var not in data_original.columns for var in X_vars):
raise ValueError("Erreur : Assurez-vous que toutes les variables existent dans le DataFrame.")
# Définir X et y
X = data_original[X_vars] # Suppression de l'ajout de la constante
y = data_original[y_var]
# Vérifier si X contient au moins une variable
if X.shape[1] == 0:
raise ValueError("Erreur : Aucune variable explicative valide sélectionnée.")
# Ajustement du modèle OLS
model = sm.OLS(y, X).fit()
print(model.summary())
# Vérification de la normalité des résidus avec un QQ-plot
residuals = model.resid
fig, ax = plt.subplots(figsize=(6, 6))
sm.qqplot(residuals, line="s", ax=ax)
ax.set_title("QQ-Plot des résidus")
plt.show()
# Vérification de l'hétéroscédasticité avec un scatter plot
plt.figure(figsize=(8, 5))
sns.scatterplot(x=model.fittedvalues, y=residuals)
plt.axhline(0, linestyle="dashed", color="red")
plt.xlabel("Valeurs ajustées")
plt.ylabel("Résidus")
plt.title("Graphique des résidus")
plt.show()
# Calcul du VIF pour détecter la multicolinéarité
if X.shape[1] > 1: # Vérifier que le VIF peut être calculé
vif_data = pd.DataFrame({
"Variable": X.columns,
"VIF": [variance_inflation_factor(X.values, i) for i in range(X.shape[1])]
})
print("\n📊 Facteur d'inflation de la variance (VIF) :")
display(vif_data)
# Interprétation du VIF
high_vif = vif_data[vif_data["VIF"] > 10]
if not high_vif.empty:
print("\n⚠️ Attention : Certaines variables présentent une forte multicolinéarité (VIF > 10). Cela peut affecter la fiabilité des coefficients estimés.")
print(high_vif)
else:
print("✅ Aucune multicolinéarité préoccupante détectée (VIF < 10).")
else:
print("\n⚠️ Pas assez de variables pour calculer le VIF.")
# Test de normalité des résidus (Jarque-Bera)
jb_stat, jb_pvalue, skew, kurtosis = sm.stats.jarque_bera(residuals)
print("\n📊 Résultats du Test de normalité de Jarque-Bera :")
print(f" - Statistique JB : {jb_stat:.3f}")
print(f" - p-value : {jb_pvalue:.5f}")
print(f" - Asymétrie (Skewness) : {skew:.3f}")
print(f" - Aplatissement (Kurtosis) : {kurtosis:.3f}")
# Interprétation automatique
test_result = "✅ Les résidus suivent une loi normale (p > 0.05)." if jb_pvalue > 0.05 else "❌ Les résidus ne suivent PAS une loi normale (p ≤ 0.05)."
print(f"\n{test_result}")
if jb_pvalue <= 0.05:
if abs(skew) > 1:
print("🔍 Problème détecté : **Asymétrie élevée** (skewness).")
print("✅ Solution : Essayez une transformation logarithmique ou une normalisation.")
if kurtosis < 2 or kurtosis > 4:
print("🔍 Problème détecté : **Aplatissement anormal (kurtosis).**")
print("✅ Solution : Vérifiez s'il y a des valeurs aberrantes (outliers).")
print("\n📌 Recommandations supplémentaires :")
print(" - Vérifiez la présence de valeurs aberrantes avec un boxplot.")
print(" - Testez une transformation (log, carré, racine) sur la variable dépendante.")
print(" - Ajoutez éventuellement d'autres variables explicatives.")
# Conclusion générale
print("\n📌 **Conclusion** :")
print(" - L'ajustement du modèle est évalué avec le R² et le R² ajusté.")
print(" - Un R² élevé (>0.7) indique une bonne capacité explicative du modèle, tandis qu'un R² faible suggère un ajustement limité.")
print(" - Les coefficients et leurs p-values indiquent la significativité des variables explicatives : une p-value < 0.05 signifie que la variable a un impact significatif.")
print(" - Le VIF aide à détecter la multicolinéarité, qui peut biaiser les estimations.")
print(" - Le test de Jarque-Bera vérifie la normalité des résidus, influençant la validité des tests de significativité.")